Recherche
- Identification de marqueurs épigénétiques spécifiques des facteurs intervenant en début de vie et associés au risque de cancer chez l’enfant (EpiEARLY)
- Identification des biomarqueurs épigénétiques de l’exposition des populations humaines associée au risque de cancer et évaluation de leur validité dans des études d’intervention (EpiMARKS)
- Identification des gènes épigénétiques « moteurs » du développement du cancer et caractérisation de leur lien avec l’exposition à des agents cancérogènes environnementaux (EpiDRIVERS)
- Etude des spectres de mutations et de leur impact, étude des marqueurs moléculaires d’exposition et du stade précoce de la maladie, et détection précoce dans des modèles expérimentaux et chez l’homme (MutSpec 2.0)
- Recherche des causes de cancer grâce aux études intégratives d’épidémiologie moléculaire des cancers
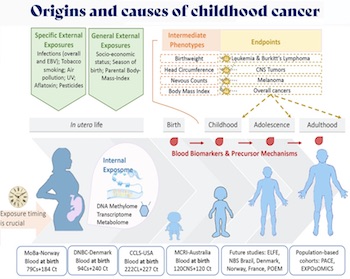
- Ce projet consiste à i) étudier les modifications épigénétiques induites par les facteurs de risque pour comprendre les mécanismes moléculaires sous-jacents aux cancers survenant chez les enfants et les adolescents et ii) identifier des marqueurs épigénétiques permettant la stratification du risque et la détection précoce du cancer. Il part de l’hypothèse qu’il est possible d’identifier à la naissance (dans les cellules sanguines) les modifications épigéniques associées au risque de cancer chez l’enfant, induites pendant la grossesse par l’exposition à des facteurs environnementaux, alimentaires et comportementaux, et que ces changements de méthylation peuvent servir de biomarqueurs spécifiques pour la prévention primaire et secondaire et la thérapie ciblée des cancers pédiatriques.
- Ce projet s’appuie sur les récentes avancées de la Branche EGM concernant l’identification de marqueurs épigénétiques des facteurs intervenant en début de vie et le décryptage de leurs rôles de précurseurs dans les cancers pédiatriques et les phénotypes (intermédiaires) prédisposant au cancer à des âges critiques du développement. Il fait appel aux larges réseaux de la Branche EGM, réseaux uniques et complémentaires de consortiums menant des études prospectives et rétrospectives (Figure 5).
- L’objectif principal de ce projet (EpiMARKS) consiste à vérifier l’hypothèse selon laquelle des changements épigénétiques particuliers peuvent être associés à et/ou causés par des facteurs alimentaires ou comportementaux spécifiques, et s’il est possible d’utiliser ces profils épigénétiques comme biomarqueurs spécifiques du risque de cancer et de l’impact des interventions préventives (prévention primaire et tertiaire) (Figure 6).
Decouverte de biomarqueurs (epi)genomiques specifiques des differents stades de developpement du cancer
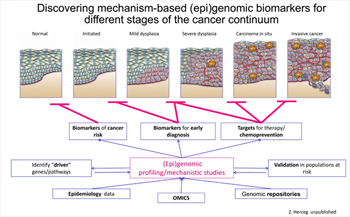
Zoom sur la Figure 6
- Les recherches portent principalement sur le cancer du sein et le cancer colorectal, deux types de cancers courants dont les principaux facteurs de risque sont identiques et modifiables (mode de vie, obésité, consommation d’alcool et sédentarité). Les taux d’incidence de ces cancers sont en hausse dans les pays à revenu faible et intermédiaire. Cette hausse peut être associée à des facteurs de risque particuliers (changements de mode de vie, exposition à des perturbateurs endocriniens) dont la présence est de plus en plus forte dans ces régions. Le projet comprend deux objectifs principaux en ce qui concerne ces deux types de cancer : i) identifier les biomarqueurs épigénétiques de l’exposition et du risque de cancer (EpiSIGNATURES), et ii) examiner l’impact des interventions sur les marqueurs épigénétiques associés au risque de cancer (EpiTRIALS). La Branche EGM s’appuie à la fois sur des cohortes prospectives uniques (qui permettent de mesurer les événements épigénétiques à différents moments avant le diagnostic, généralement dans des tissus de substitution tels que le sang), des études cas-témoins (qui couvrent les régions à forte incidence et permettent de détecter les aberrations épigénétiques directement dans les tissus constituant des « cibles tumorales ») et des études d’intervention (qui permettent d’examiner la possibilité d’interventions ciblées visant à inverser les schémas épigénétiques associés à la maladie et à déchiffrer les associations « causales » par expérimentation directe).
- Un autre axe de recherche dans ce domaine consiste à identifier les marqueurs épigénétiques du carcinome épidermoïde œsophagien en se focalisant sur les populations à forte incidence (EpiESCC). Ce projet vise à comprendre les causes biologiques sous-jacentes au carcinome épidermoïde œsophagien dans ces populations en étudiant les aberrations épigénétiques (méthylation de l’ADN) induites par les facteurs de risque, et en identifiant des marqueurs moléculaires permettant la stratification du risque et la détection précoce du cancer. Ces recherches s’appuient sur l’étude épidémiologique unique, coordonnée par le CIRC, du carcinome épidermoïde de l’œsophage dans les populations à forte incidence, qui a permis de collecter de nombreux échantillons de tumeurs, de tissus normaux adjacents aux tumeurs et de tissus de substitution (cellules sanguines), ainsi que des données épidémiologiques et cliniques de haute qualité.
- La fréquence élevée des altérations des gènes codants pour les protéines qui régulent directement l’épigénome – appelés ici gènes régulateurs épigénétiques (GREs) – dans les cancers humains les plus courants, constitue l’un des résultats les plus remarquables des études internationales de séquençage à haute résolution du génome du cancer, dont l’Atlas du génome du cancer (TCGA pour The Cancer Genome Atlas) est le fer de lance. Ce taux élevé de dérégulation de ces gènes régulateurs constitue une « preuve génétique irréfutable » que les mécanismes épigénétiques sont au cœur même de la biologie du cancer. On ignore toutefois encore beaucoup de choses concernant l’impact de ces altérations sur l’expression et la fonction des GREs dans les cancers. Ces résultats ont suscité un vif débat sur le rôle « moteur » qu’ils pourraient jouer dans la tumorigenèse et dans les mécanismes sous-jacents aux modifications épigéniques observées dans les tumeurs malignes humaines. Cette étude (EpiDRIVERS) vise à tester l’hypothèse selon laquelle les GREs, lorsqu’ils sont perturbés par des mécanismes génétiques ou épigénétiques (mécanismes ne faisant pas intervenir des altérations de la séquence de l’ADN), peuvent agir comme des gènes moteurs (« epidrivers ») dans le développement du cancer et conférer un phénotype de cellule cancéreuse (éventuellement en synergie avec une exposition environnementale) (Figure 7). Ce projet s’appuie sur les travaux antérieurs de la Branche EGM à l’origine de découvertes importantes concernant le rôle des régulateurs épigénétiques dans des processus chromatiniens, dont la transcription et la réparation de l’ADN.
- Récemment, la Branche EGM a mis au point et appliqué de nouvelles approches bioinformatiques et statistiques, intégrant divers algorithmes permettant de prédire quels sont les gènes moteurs. Ces approches ont ainsi permis d’identifier des GREs jouant un rôle moteur dans des tumeurs malignes et entre des tumeurs malignes. La Branche EGM a également développé et testé un cadre conceptuel pour identifier et valider dans des modèles cellulaires tridimensionnels, les gènes « epidrivers » fonctionnellement importants, par le biais d’une nouvelle approche systématique qui intègre les capacités de criblage d’édition du génome (CRISPR/Cas9). Les résultats de ce criblage ont permis d’identifier un panel de gènes régulateurs susceptibles de jouer le rôle moteur conférant aux cellules cancéreuses les traits associés aux caractéristiques spécifiques (phénotype) du cancer. D’autres études s’attachent à caractériser les gènes « epidrivers » qui jouent un rôle essentiel dans la tumorigenèse et à examiner le rôle que pourraient jouer certains d’entre eux dans l’association entre expositions environnementales et cancer, notamment avec l’accumulation de plusieurs traits phénotypiques tout au long du processus de cancérogenèse et l’implication de différents systèmes biologiques. La réponse à cette question devrait permettre de mieux comprendre les mécanismes de cancérogenèse, faciliter la classification des tumeurs et fournir des informations importantes pour l’évaluation et la classification des agents cancérogènes.
Cadre conceptuel pour l’analyse multi-omique du cancer et l’evaluation experimentale des genes regulateurs des processus epigenetiques (« genes epidrivers ») et de leur lien avec les agents cancerogenes environnementaux
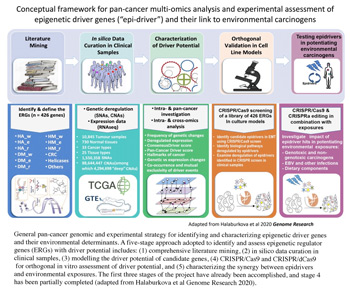
Zoom sur la Figure 7
© 2020 Halaburkova et coll.; Cold Spring Harbor Laboratory Press.
Stratégie d’étude expérimentale et pangénomique du cancer pour l’identification et la caractérisation des gènes « epidrivers » et de leurs déterminants environnementaux. Une stratégie en cinq étapes a été adoptée pour identifier et évaluer les gènes régulateurs des processus épigénétiques (GREs) pouvant être des gènes moteurs : 1) exploration intensive de la littérature ; 2) conservation in silico des données des échantillons cliniques ; 3) modélisation du potentiel « moteur » des gènes candidats, 4) utilisation de CRISPR/Cas9 et CRISPR/dCas9 pour l’évaluation orthogonale in vitro du potentiel « moteur », et 5) caractérisation de la synergie entre « epidrivers » et expositions environnementales. Les trois premières étapes du projet ont déjà été réalisées et la phase 4 est partiellement achevée (adapté de Halaburkova et coll, Genome Research 2020).
- Parmi les principales activités de recherche figurent les études toxicogénomiques expérimentales des agents potentiellement cancérogènes (projet MutSpec 2.0). Il s’agit de caractériser de façon méthodique, dans des modèles cellulaires, les effets de l’exposition aux agents cancérogènes sur le génome ; d’analyser le spectre des mutations présentes dans des tissus animaux (provenant d’essais biologiques) et des tumeurs d’origine humaine ; et de mener des études collaboratives pour identifier les agents présentant un risque de cancer sur lesquels il conviendra de réaliser en priorité les tests de mutagénicité (Figure 8).
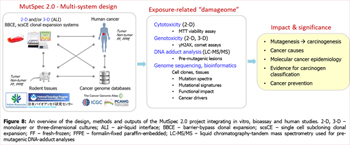
Zoom sur la Figure 8
Aperçu de la conception, des méthodes et des résultats du projet MutSpec 2.0 intégrant des études in vitro, des essais biologiques et des études chez les humains. Cultures cellulaires 2D et 3D en monocouches ou tridimensionnelles ; ALI – interface air-liquide ; BBCE – expansion clonale par contournement de barrière ; scsCE – expansion clonale par sous-clonage d’une seule cellule ; FF – congelé frais ; FFPE – fixé au formol et inclus dans la paraffine ; LC-MS/MS – utilisation de la chromatographie liquide couplée à la spectrométrie de masse pour analyser les adduits à l’ADN pré-mutagènes.
- Le projet MutSpec 2.0 fait appel à des méthodes expérimentales et à des systèmes innovants, conçus spécialement dans ce cadre. A ce jour, le projet s’attache à perfectionner les systèmes modèles existants pour i) mettre au point des applications à haut débit permettant de tester les agents potentiellement cancérogènes, et ii) à étudier les mécanismes qui endommagent l’ADN, en s’appuyant notamment sur des études collaboratives pour analyser les adduits à l’ADN (approches adductomiques) et identifier ainsi des lésions pré-mutagènes. L’impact fonctionnel des agressions mutagènes est étudié à travers l’identification des relations entre mutagenèse et topographie du génome, et l’analyse des spectres de mutation et de la dynamique de l’organisation de la chromatine pendant l’immortalisation et la transformation des cellules et dans la tumeur. L’accent est mis sur l’interaction fonctionnelle entre les altérations génétiques et épigénétiques au cours des premières étapes de la transformation cellulaire, ainsi que sur la validation de marqueurs moléculaires expérimentaux issus de données publiques sur le cancer.
- Forte de son expertise en génomique et en bioinformatique concernant les agressions génotoxiques par des cancérogènes présumés ou connus, la Branche EGM participe à diverses études d’épidémiologie moléculaire du cancer, notamment (mais pas exclusivement) aux enquêtes sur la cancérogénicité rénale liée à l’exposition alimentaire à l’acrylamide (projet MODARC, soutenu par le World Cancer Research Fund International), le rôle de l’exposition à l’amiante dans le développement du cancer de l’ovaire (étude EVAMOVAIRE2, soutenue par l’ANSES, France), les signatures métaboliques et génétiques dans le cancer du sein triple négatif de la cohorte EPIC (soutenue par l’INCa, France), et le risque de cancer associé à l’exposition à l’acide aristolochique, agent cancérogène extrait de plantes, en Asie et en Europe. Dans les pays à revenu faible et intermédiaire, la Branche EGM collabore à l’étude des bases moléculaires des cancers à forte incidence dans certaines régions (projet ARISTOCANCERS). Les études mécanistiques et d’épidémiologie moléculaire utilisant des modèles permettent d’identifier des biomarqueurs d’exposition et de développement du cancer, qui devraient pouvoir s’appliquer à l’évaluation des dangers cancérogènes.